Ejemplo USairpollution
Los paquetes de R que vamos a emplear son:
# Lectura de datos
library(readr)
# Manipulación de datos
library(tidyverse)
library(magrittr)
# Gráficos
library(ggplot2)
library(ggforce)
library(ggthemes)Los datos se pueden encontar en el paquete HSAUR3.
data("USairpollution", package="HSAUR3")También se pueden cargar desde el directorio de trabajo.
aire.dat <- read_table2("Datos/datosaire.txt")
aire.dat %>% head()# A tibble: 6 x 8
Ciudad SO2 Temp Empresas Pob Viento Precip Dias
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Phoenix 10 70.3 213 582 6 7.05 36
2 LittleRock 13 61 91 132 8.2 48.5 100
3 SanFrancisco 12 56.7 453 716 8.7 20.7 67
4 Denver 17 51.9 454 515 9 13.0 86
5 Hartford 56 49.1 412 158 9 43.4 127
6 Wilmington 36 54 80 80 9 40.2 114Las variables recogidas en la base de datos son las siguientes:
Ciudad: nombre de la ciudad de EE.UU.SO2: contenido de SO2 del aire en microgramos por metro cúbico.Temp: temperatura media anual en grados Fahrenheit.Empresas: número de empresas manufactureras que emplean 20 o más trabajadores.Pob: tamaño de la población (censo de 1970) en miles.Viento: velocidad media anual del viento en millas por hora.Precip: precipitación media anual en pulgadas.Dias: número medio de días con precipitación al año
Guardamos la variable Ciudad como nombres de la matriz y la quitamos para tener una matriz de datos numéricos. Es mejor usar el formato tibble en los datos que el dataframe por cuestiones de uso del paquete tidyverse, no obstante está modificación no es estrictamente necesaria para el buen desarrollo del ejercicio aunque la efectuaremos.
aire.dat <- aire.dat %>% as.tibble() %>% column_to_rownames("Ciudad")Podemos realizar un estudio descriptivo de los datos para observar la singularidad de estos y advertir posibles anomalías. Además, complementaremos el análisis descriptivo con gráficos ilustrativos usando el paquete ggplot2, para lo que necesitaremos a menudo realizar una transformación, pivotado, de los datos para adecuarlos a la sintaxis del paquete e incluso emplear el paquete forcast para mejorar la visualización de los gráficos.
- Análisis descriptivo.
aire.dat %>% summary() SO2 Temp Empresas Pob
Min. : 8.00 Min. :43.50 Min. : 35.0 Min. : 71.0
1st Qu.: 13.00 1st Qu.:50.60 1st Qu.: 181.0 1st Qu.: 299.0
Median : 26.00 Median :54.60 Median : 347.0 Median : 515.0
Mean : 30.05 Mean :55.76 Mean : 463.1 Mean : 608.6
3rd Qu.: 35.00 3rd Qu.:59.30 3rd Qu.: 462.0 3rd Qu.: 717.0
Max. :110.00 Max. :75.50 Max. :3344.0 Max. :3369.0
Viento Precip Dias
Min. : 6.000 Min. : 7.05 Min. : 36.0
1st Qu.: 8.700 1st Qu.:30.96 1st Qu.:103.0
Median : 9.300 Median :38.74 Median :115.0
Mean : 9.444 Mean :36.77 Mean :113.9
3rd Qu.:10.600 3rd Qu.:43.11 3rd Qu.:128.0
Max. :12.700 Max. :59.80 Max. :166.0 - Diagrama de cajas.
aire.dat %>%
pivot_longer(everything(),names_to = "item",values_to = "valor") %>%
mutate(item = fct_reorder(item, valor, .fun='median')) %>%
ggplot(aes(x=item, y=valor, fill=item)) +
geom_boxplot() + theme_pander() +
theme(legend.position = "none") +
xlab("") + ylab("")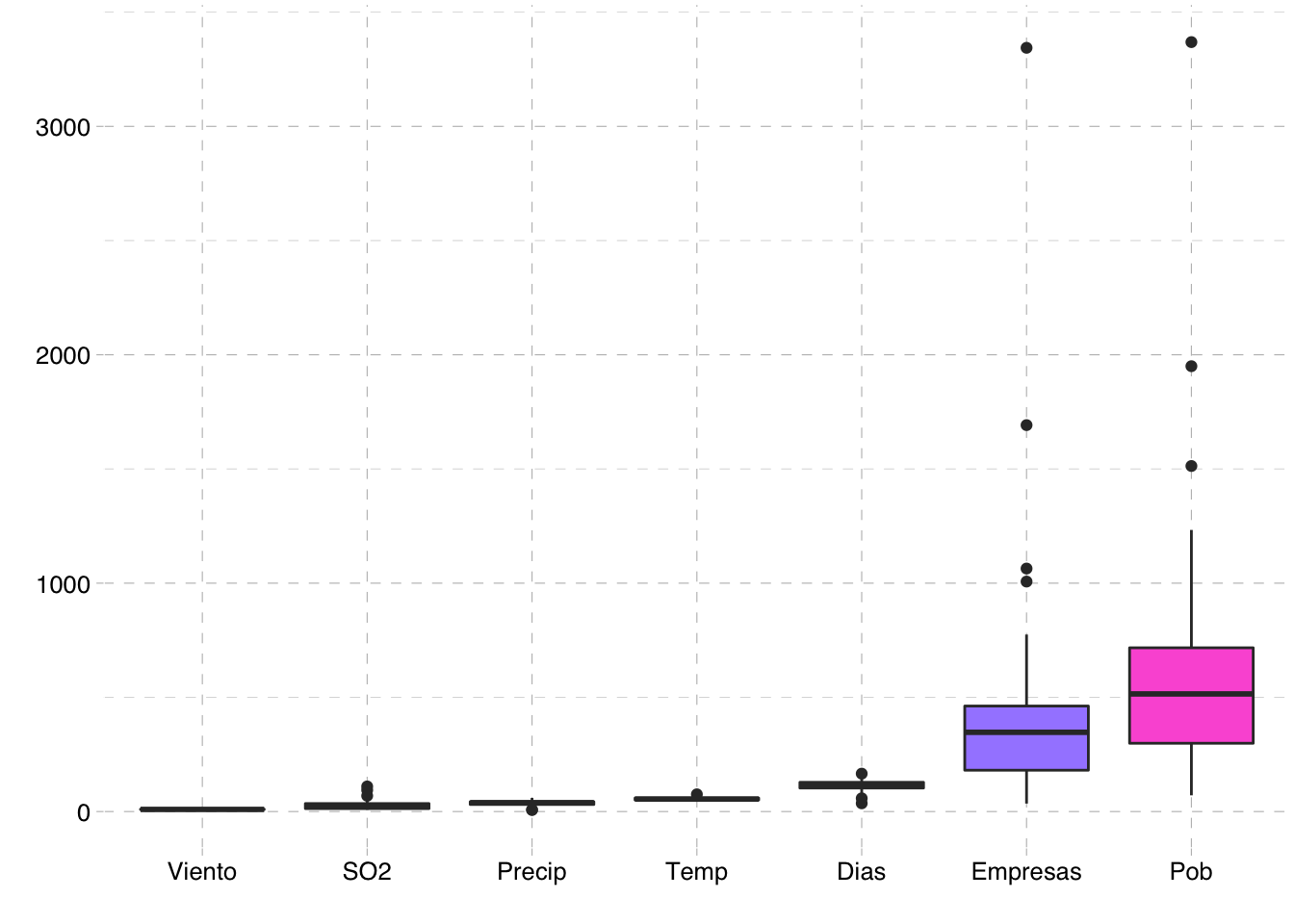
- Gráfico de dispersión de parejas de variables. Se puede hacer llamando a la función
pairs()implementada en R base pero para seguir con la sintaxis del paqueteggplot2encontramos una opción en el paqueteGGallyque proporciona mucha más información con menor código.
library(GGally)
aire.dat %>%
ggpairs() + theme_pander() +
theme(axis.text.x = element_text(angle = 90, vjust = 0.5, hjust=1))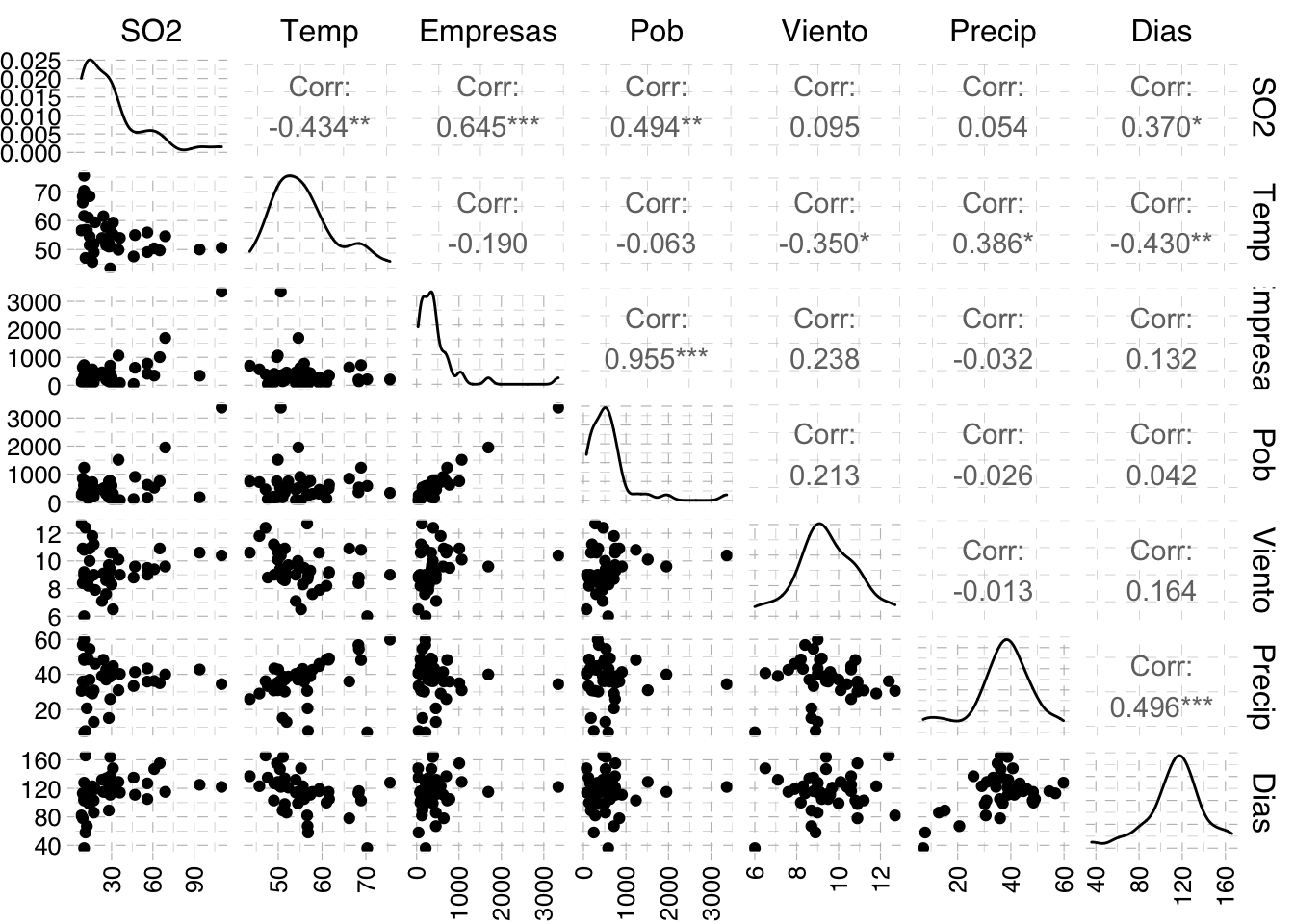
Los gráficos sugieren que pueden existir algunos valores atípicos en nuestra muestra. Por otra parte, en el diagrama de cajas podemos observar que las variables se miden en escalas bastante diferentes (unidades y rango de valores bastante diferentes), por lo que parece mejor trabajar con las variables estandarizadas. Además, encontramos que existen relaciones de colinealidad entre variables como por ejemplo entre Empresas y Pob. De hecho, como se comentaba en el desarrollo teórico anterior, para poder deducir conclusiones válidas del ACP es fundamental que exista correlación entre variables. Veamos si se cumple estudiando el determinante de la matriz de correlaciones.
- Estudio de la correlación. Vamos a calcular la matriz de correlaciones, proyectar una visualización de esta reccurriendo al
heatmap(se usará el paquetecorrplot) y computar el determinante de la matriz.
R=cor(aire.dat)
round(R,2) SO2 Temp Empresas Pob Viento Precip Dias
SO2 1.00 -0.43 0.64 0.49 0.09 0.05 0.37
Temp -0.43 1.00 -0.19 -0.06 -0.35 0.39 -0.43
Empresas 0.64 -0.19 1.00 0.96 0.24 -0.03 0.13
Pob 0.49 -0.06 0.96 1.00 0.21 -0.03 0.04
Viento 0.09 -0.35 0.24 0.21 1.00 -0.01 0.16
Precip 0.05 0.39 -0.03 -0.03 -0.01 1.00 0.50
Dias 0.37 -0.43 0.13 0.04 0.16 0.50 1.00library(corrplot)
R %>% corrplot(method = "square")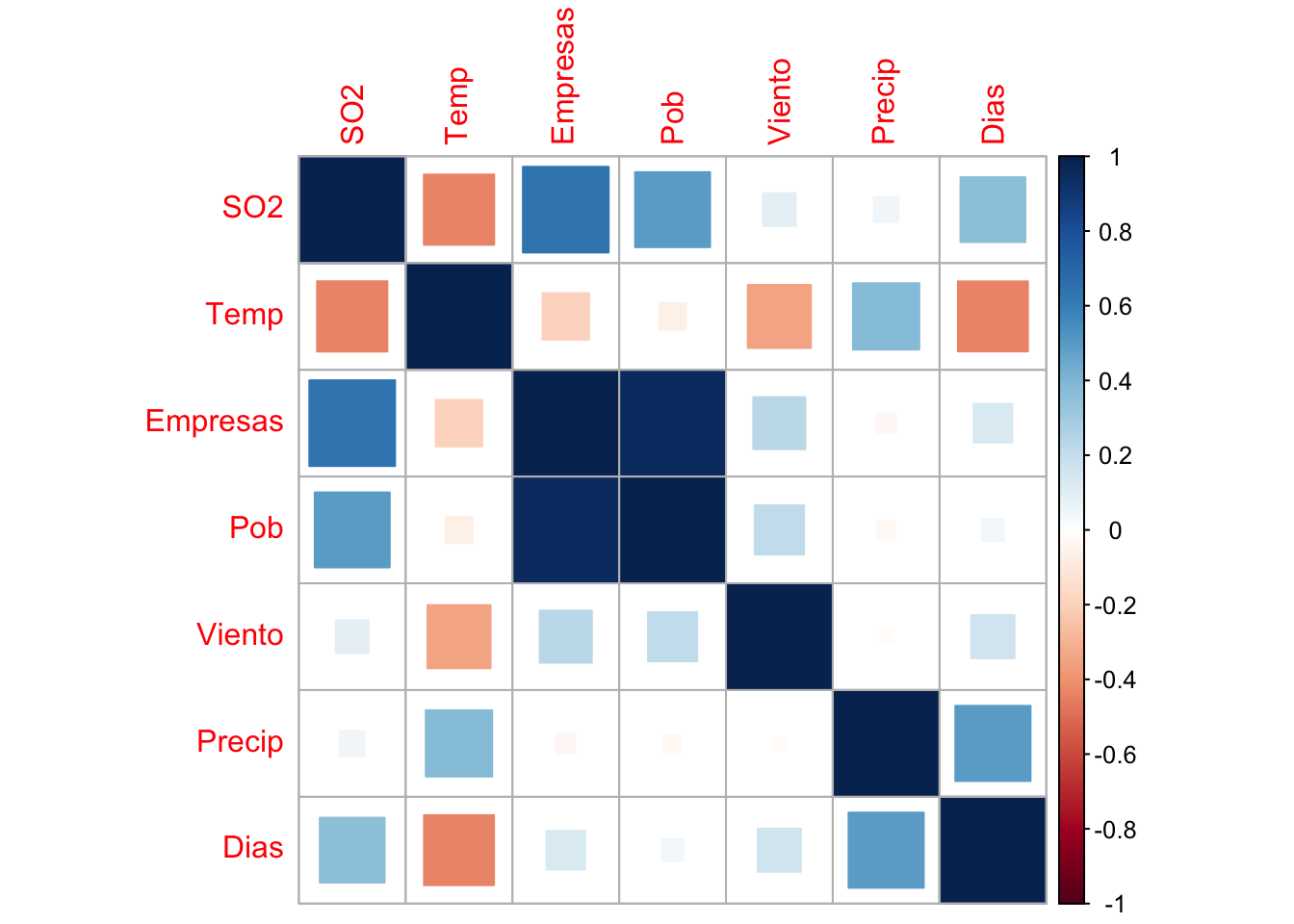
det(R)[1] 0.004555454El determinante de la matriz de correlaciones: un determinante muy bajo indicará altas intercorrelaciones entre las variables, pero no debe ser cero (matriz no singular), pues esto indicaría que algunas de las variables son linealmente dependientes y no se podrían realizar ciertos cálculos.
En nuestra matriz, dado que det(R) está cerca de cero, existe colinealidad y el ACP es apropiado para tratar con este conjunto de datos.
ACP con princomp
ORDEN:
princomp(formula, data = NULL, subset, na.action, ...)princomp(x, cor = FALSE, scores = TRUE, covmat = NULL, subset = rep(TRUE, nrow(as.matrix(x))), ...)
ARGUMENTOS:
formula: fórmula sin variable respuesta, sólo con variables numéricas. Por ej.:~ varX1 + varX2 + varX3.data: un marco de datos opcional que contenga las variables de la fórmula.subset: un vector opcional para seleccionar las filas (observaciones) de la matriz de datos.x: matriz o marco de datos que proporciona los datos que proporciona los datos para el ACP.cor: Valor lógico (T/F) indicando si se usa la matriz de correlación (T) o la matriz de covarianzas (F). Dado que debemos considerar las variables estandarizadas, o equivalentemente la matriz de correlación,cor = TRUE.scores: Valor lógico (T/F) indicando si las puntuaciones de cada c.p. deben ser calculadas.
RESULTADOS: Crea un objeto princomp que recoge la siguiente información:
sdev: desviaciones estándar de las comp.principales.loadings: matriz de cargas (es decir, matriz de autovectores).center: las medias.scaling: la escala aplicada a cada variable.n.obs: número de observaciones.scores: Si se ha solicitado, las puntuaciones de los datos en las c.p.
pca <- princomp(aire.dat, cor = TRUE)
pcaCall:
princomp(x = aire.dat, cor = TRUE)
Standard deviations:
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
1.6517021 1.2297702 1.1810897 0.9444529 0.5888792 0.3166822 0.1597339
7 variables and 41 observations.pca %>% str()List of 7
$ sdev : Named num [1:7] 1.652 1.23 1.181 0.944 0.589 ...
..- attr(*, "names")= chr [1:7] "Comp.1" "Comp.2" "Comp.3" "Comp.4" ...
$ loadings: 'loadings' num [1:7, 1:7] 0.49 -0.315 0.541 0.488 0.25 ...
..- attr(*, "dimnames")=List of 2
.. ..$ : chr [1:7] "SO2" "Temp" "Empresas" "Pob" ...
.. ..$ : chr [1:7] "Comp.1" "Comp.2" "Comp.3" "Comp.4" ...
$ center : Named num [1:7] 30.05 55.76 463.1 608.61 9.44 ...
..- attr(*, "names")= chr [1:7] "SO2" "Temp" "Empresas" "Pob" ...
$ scale : Named num [1:7] 23.18 7.14 556.56 572.01 1.41 ...
..- attr(*, "names")= chr [1:7] "SO2" "Temp" "Empresas" "Pob" ...
$ n.obs : int 41
$ scores : num [1:41, 1:7] -2.716 -1.718 -0.939 -0.55 0.46 ...
..- attr(*, "dimnames")=List of 2
.. ..$ : chr [1:41] "Phoenix" "LittleRock" "SanFrancisco" "Denver" ...
.. ..$ : chr [1:7] "Comp.1" "Comp.2" "Comp.3" "Comp.4" ...
$ call : language princomp(x = aire.dat, cor = TRUE)
- attr(*, "class")= chr "princomp"pca %>% summary()Importance of components:
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5
Standard deviation 1.6517021 1.2297702 1.1810897 0.9444529 0.58887916
Proportion of Variance 0.3897314 0.2160478 0.1992819 0.1274273 0.04953981
Cumulative Proportion 0.3897314 0.6057792 0.8050611 0.9324884 0.98202821
Comp.6 Comp.7
Standard deviation 0.3166822 0.159733920
Proportion of Variance 0.0143268 0.003644989
Cumulative Proportion 0.9963550 1.000000000La función de resumen en el objeto de resultado nos da la desviación estándar, la proporción de varianza explicada por cada componente principal y la proporción acumulada de varianza explicada. Recuérdese que la varianza del
Podemos construir nuestro propio resumen.
resumen_name <- paste0("CP",1:7)
resumen_eign <- pca$sdev^2
resumen_CP <- tibble(CP = resumen_name, Eigen = resumen_eign) %>%
mutate(Percentage = 100*Eigen/sum(Eigen),
`Cumulative Percentage` = cumsum(Percentage))
resumen_CP %>%
mutate_at(2:4, round, 2) %>%
kable()| CP | Eigen | Percentage | Cumulative Percentage |
|---|---|---|---|
| CP1 | 2.73 | 38.97 | 38.97 |
| CP2 | 1.51 | 21.60 | 60.58 |
| CP3 | 1.39 | 19.93 | 80.51 |
| CP4 | 0.89 | 12.74 | 93.25 |
| CP5 | 0.35 | 4.95 | 98.20 |
| CP6 | 0.10 | 1.43 | 99.64 |
| CP7 | 0.03 | 0.36 | 100.00 |
Comprobamos que los valores propios de la matriz
eigen(R)$values[1] 2.72811968 1.51233485 1.39497299 0.89199129 0.34677866 0.10028759 0.02551493Selección del número de componentes
Podemos ver la variablidad explicada por cada componente construyendo un gráfico de autovalores o scree plot. Esto nos facilitará ver con qué componentes principales nos quedaremos siguiendo uno de los criterios anteriormente expuestos.
resumen_CP %>%
ggplot(aes(x = CP, y = Eigen)) +
geom_bar(stat="identity",width=0.01) +
geom_point() + theme_pander() +
geom_hline(yintercept=1, linetype="dashed", color = "red")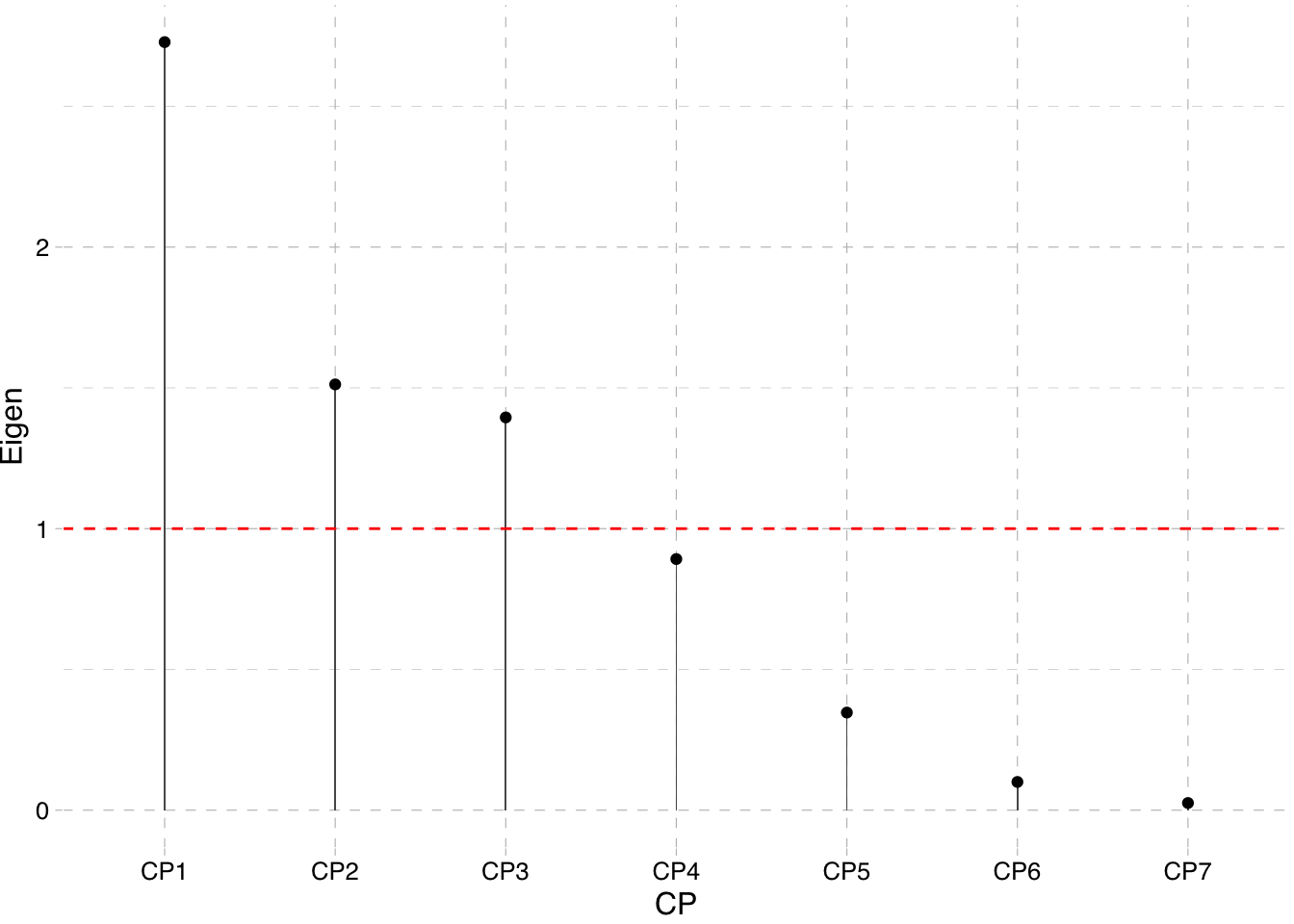
Para observar la variablidad explicada por cada CP existe una opción en el paquete de R base es llamando a plot directamente.
plot(pca, col = "lightblue")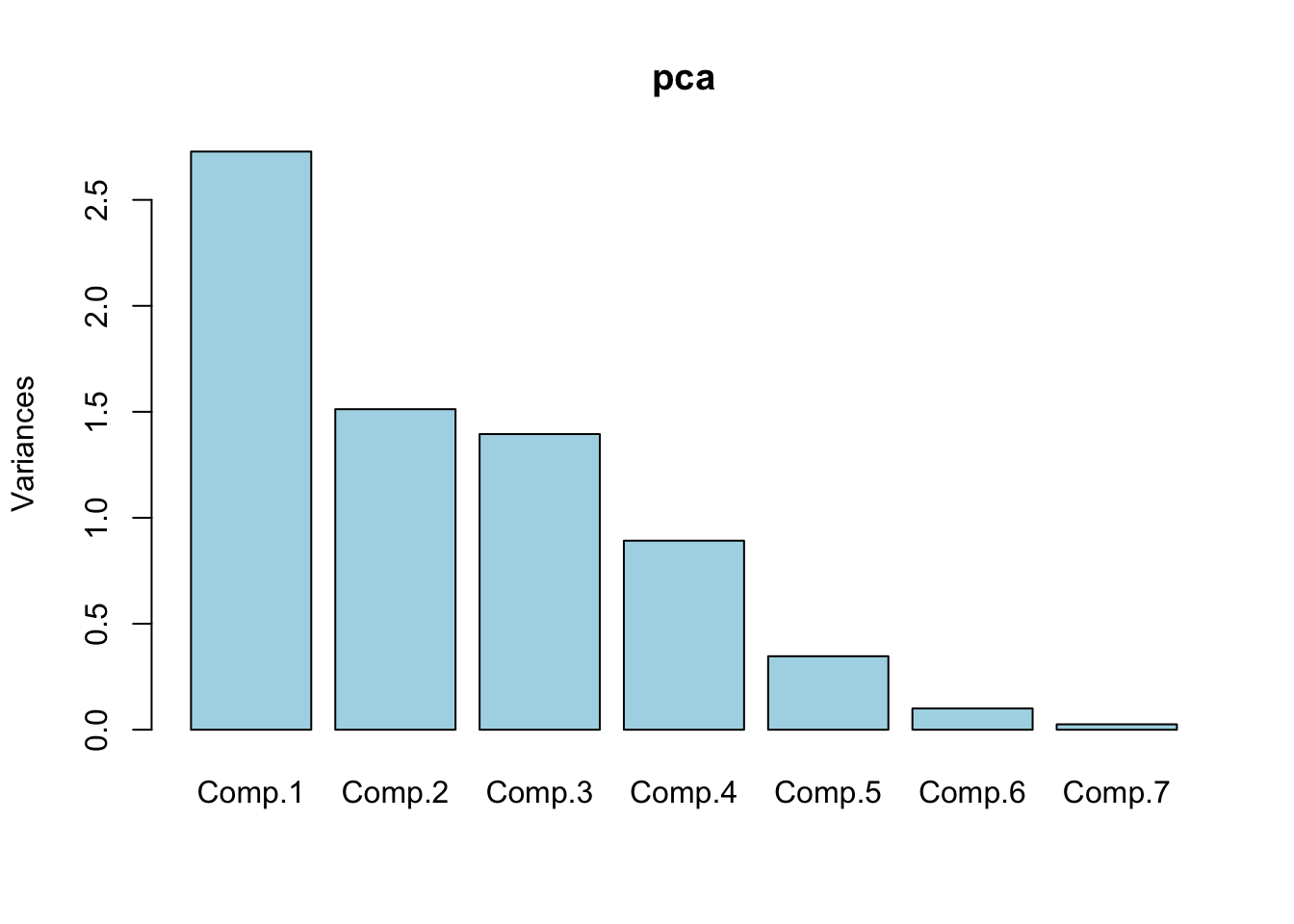
Tanto el resumen del ACP como ambos dibujos sugieren que en nuestro caso nos deberíamos de quedar con 3 componentes.
Estudio de las componentes
- Cargas (loadings) de cada CP. Las conseguimos llamando a la orden
loadingsdel paquete estadístico de R base. Las cargas pequeñas no se imprimen convencionalmente (siendo reemplazadas por espacios), para destacar los patrones de las cargas más grandes.
pca %>% loadings()
Loadings:
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
SO2 0.490 0.404 0.730 0.183 0.150
Temp -0.315 0.677 -0.185 0.162 0.611
Empresas 0.541 -0.226 0.267 -0.164 -0.745
Pob 0.488 -0.282 0.345 -0.113 -0.349 0.649
Viento 0.250 -0.311 -0.862 0.268 0.150
Precip 0.626 0.492 -0.184 0.161 -0.554
Dias 0.260 0.678 -0.110 0.110 -0.440 0.505
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
SS loadings 1.000 1.000 1.000 1.000 1.000 1.000 1.000
Proportion Var 0.143 0.143 0.143 0.143 0.143 0.143 0.143
Cumulative Var 0.143 0.286 0.429 0.571 0.714 0.857 1.000Recuérdese que las cargas son los coeficientes
y así con las demás. Las cargas coinciden con los autovectores de la matriz
eigen(R)$vectors %>%
as.tibble() %>%
select(1:3) %>%
round(3) %>%
kable()| V1 | V2 | V3 |
|---|---|---|
| 0.490 | -0.085 | -0.014 |
| -0.315 | 0.089 | -0.677 |
| 0.541 | 0.226 | -0.267 |
| 0.488 | 0.282 | -0.345 |
| 0.250 | -0.055 | 0.311 |
| 0.000 | -0.626 | -0.492 |
| 0.260 | -0.678 | 0.110 |
Coinciden salvo signo.1
- Correlación entre las variables originales
La relación de correlación viene dada por
correlations <- loadings(pca)%*%diag(pca$sdev)
corr_tibble <- correlations %>%
set_colnames(paste0("CP",1:7)) %>%
as.tibble() %>% mutate(nombre = rownames(correlations))
corr_tibble# A tibble: 7 x 8
CP1 CP2 CP3 CP4 CP5 CP6 CP7 nombre
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr>
1 0.809 0.104 0.0169 0.382 0.430 0.0581 0.0239 SO2
2 -0.521 -0.109 0.800 -0.175 0.0957 0.193 -0.00378 Temp
3 0.894 -0.278 0.316 -0.0248 -0.0966 -0.0135 -0.119 Empresas
4 0.805 -0.347 0.407 -0.107 -0.206 -0.0278 0.104 Pob
5 0.413 0.0682 -0.368 -0.814 0.158 0.0475 0.00252 Viento
6 0.000309 0.770 0.581 -0.174 0.0946 -0.175 -0.00165 Precip
7 0.430 0.834 -0.129 0.104 -0.259 0.160 0.00131 Dias corr_tibble %>%
select(c(1:2,8)) %>%
ggplot(aes(CP1,CP2,label=nombre)) +
xlim(-1,1) + ylim(-1,1) +
geom_label() +
geom_hline(yintercept = -0.5, linetype="dashed", color = "red") +
geom_hline(yintercept = 0.5, linetype="dashed", color = "red") +
geom_vline(xintercept = -0.5, linetype="dashed", color = "red") +
geom_vline(xintercept = 0.5, linetype="dashed", color = "red") +
theme_pander()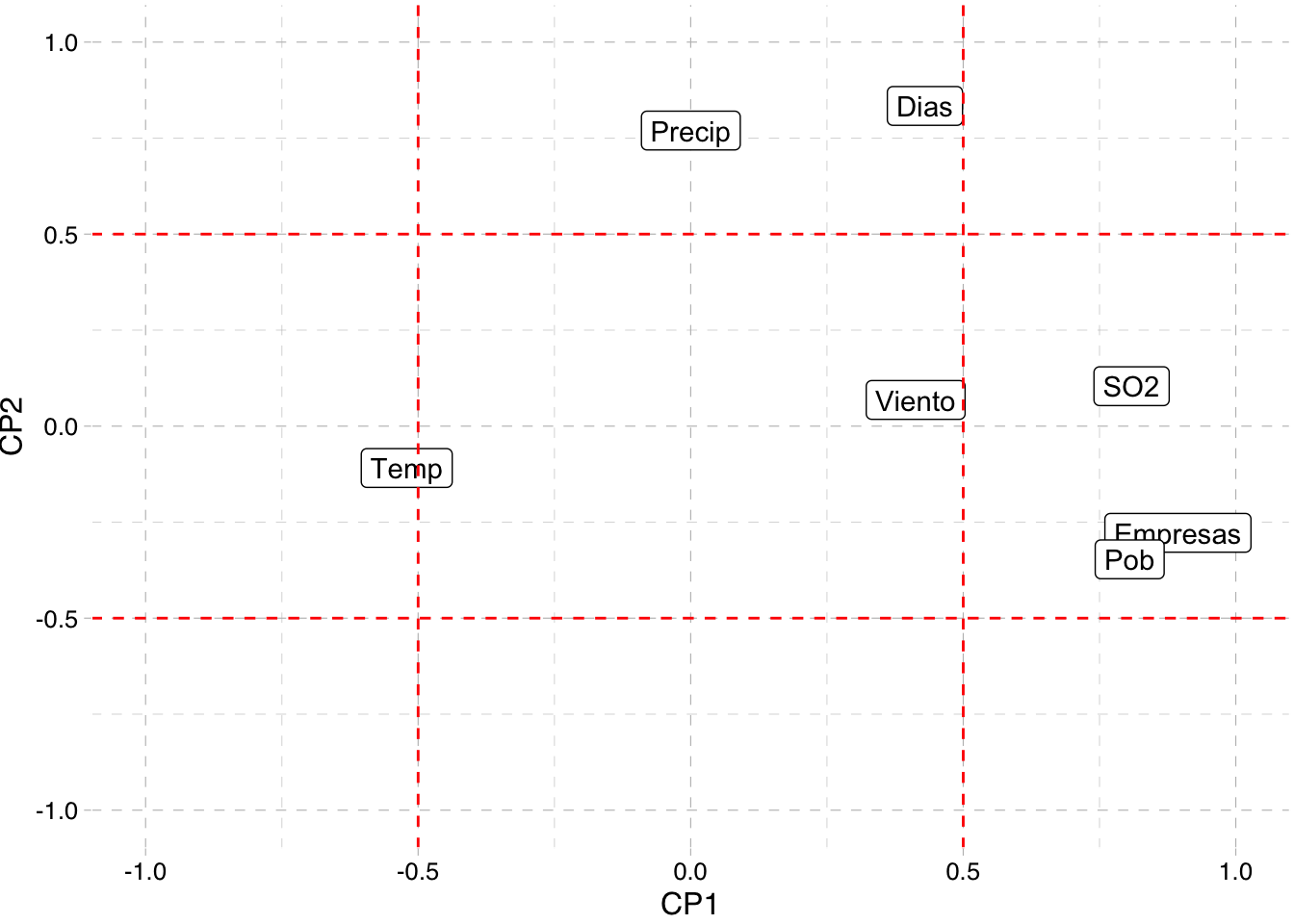
Este gráfico muestra cuáles de las variables originales están más fuertemente correlacionadas con las dos primeras CP. Podemos considerar más correladas aquellas que se encuentren fuera del cuadrado
Un gráfico al que se recurre para el estudio de la correlación entre variables y CP es el Círculo de Correlación. Se puede emplear el paquete FactoMineR como se indica en los apuntes. Otra opción es recurrir al paquete factoextra que hace un wrapper a funciones de ggplot2.
library(factoextra)
fviz_pca_var(pca, col.var = "salmon",
ggtheme = theme_pander())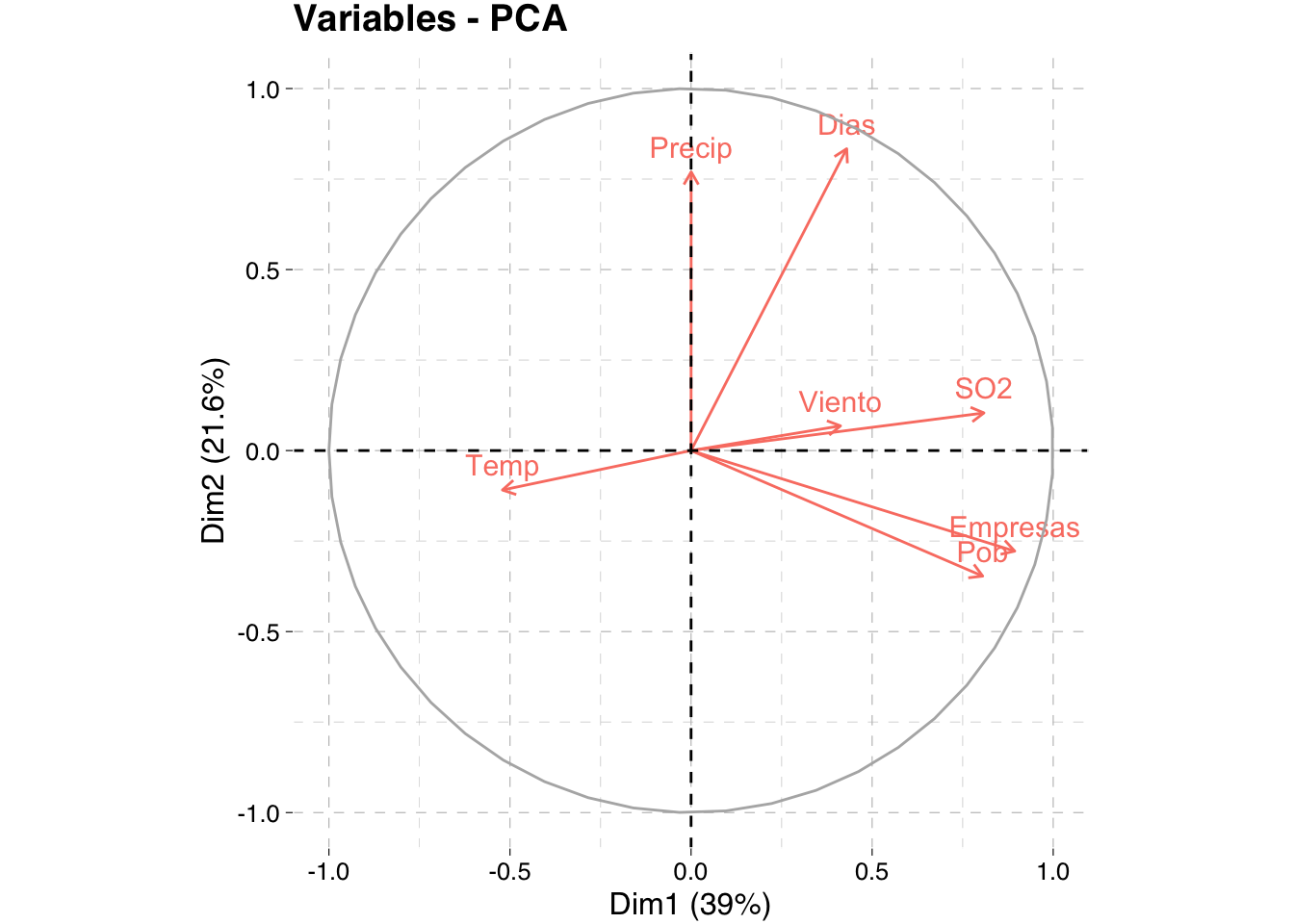
El círculo de correlación es un gráfico de
El argumento col.var puede ser una variable continua o una variable factorial. Los valores posibles incluyen: cos2, contrib, coord, x o y.
En este caso, los colores de los individuos/variables se controlan automáticamente por sus cualidades de representación (cos2), contribuciones (contrib), coordenadas (
- cos2: representa la calidad de representación de las variables y se calcula como las coordenadas cuadradas:
fviz_cos2(pca, choice = "var", axes = 1:2,
color = "salmon", fill = "cornflowerblue",
ggtheme = theme_pander())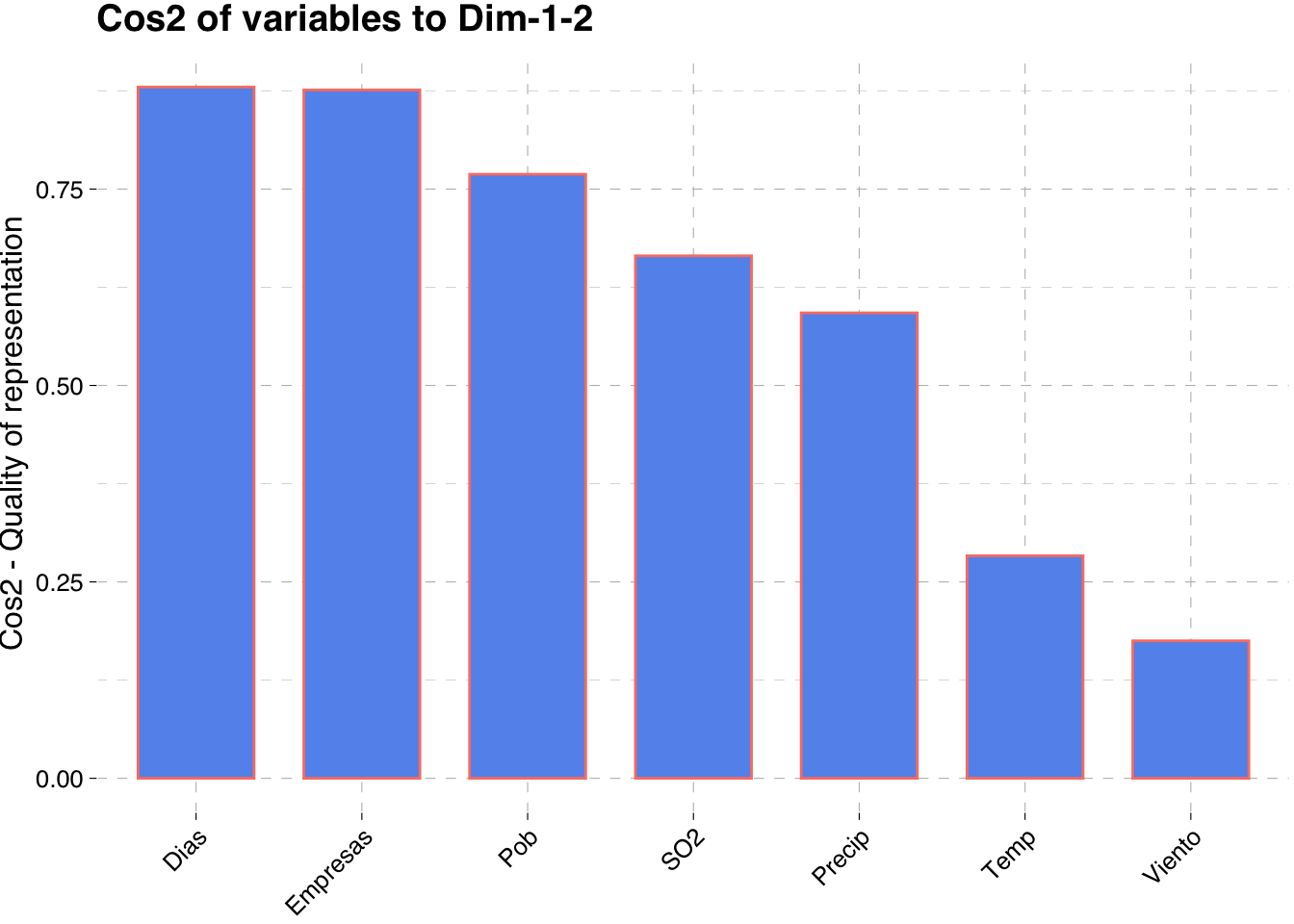
- contrib: contiene las contribuciones (en porcentaje) de las variables a los componentes principales. La contribución de una variable (var) a un componente principal dado es (en porcentaje):
Es posible utilizar la función corrplot para resaltar las variables que más contribuyen a cada CP - dimensión:
var <- get_pca_var(pca)
var$contrib %>% round(3) Dim.1 Dim.2 Dim.3 Dim.4 Dim.5 Dim.6 Dim.7
SO2 23.980 0.715 0.021 16.339 53.348 3.362 2.236
Temp 9.946 0.786 45.851 3.431 2.639 37.291 0.056
Empresas 29.286 5.102 7.137 0.069 2.693 0.183 55.529
Pob 23.774 7.953 11.891 1.286 12.187 0.772 42.136
Viento 6.244 0.308 9.689 74.287 7.196 2.252 0.025
Precip 0.000 39.172 24.210 3.383 2.579 30.644 0.011
Dias 6.769 45.964 1.201 1.205 19.357 25.497 0.007corrplot(var$contrib, is.corr=FALSE)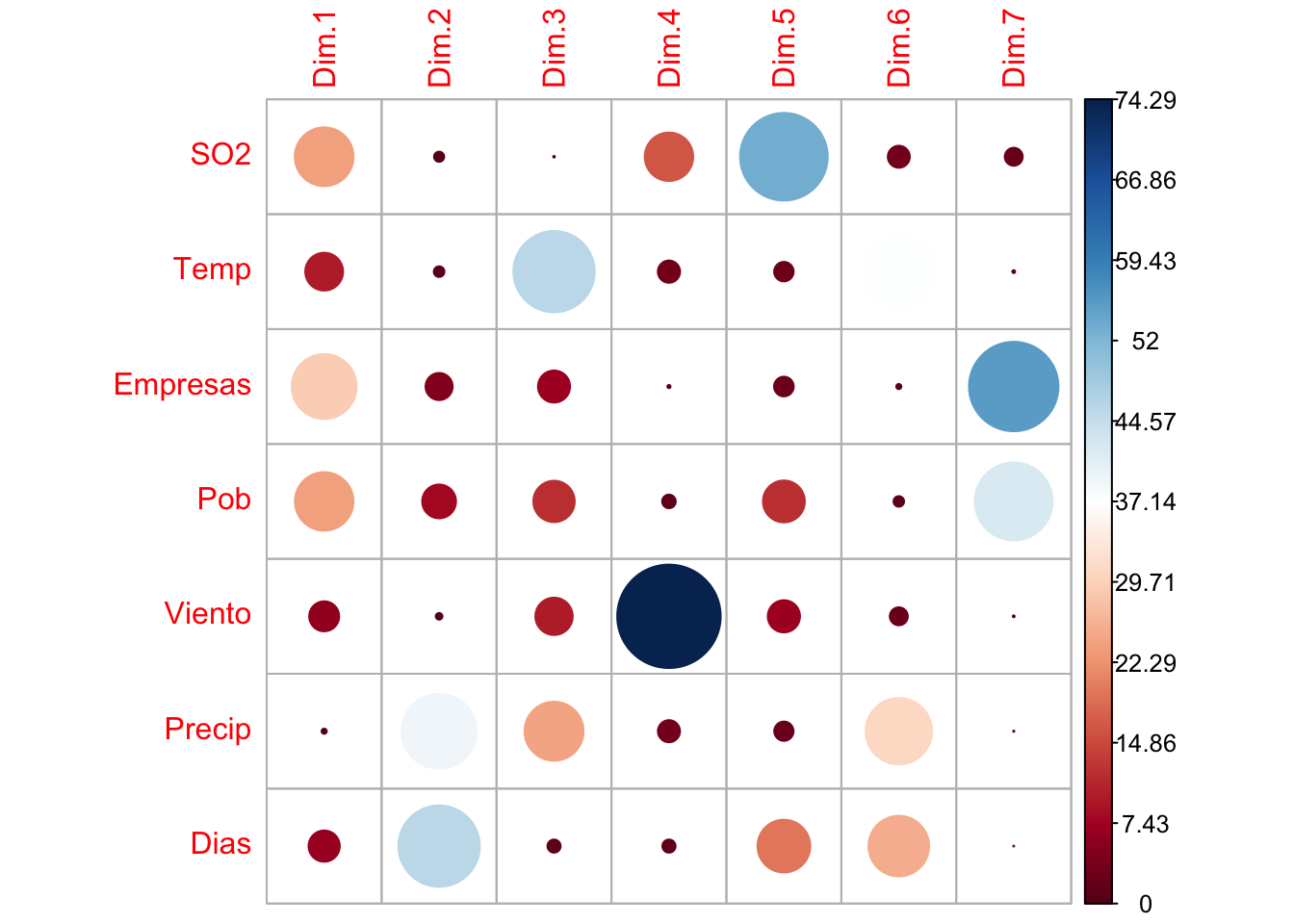
Cada cuadrado es un valor, círculos más oscuros y más grandes corresponden a valores más altos. La contribución total a CP1 y CP2 se obtiene con el siguiente código R:
fviz_contrib(pca, choice = "var", axes = 1:2, top = 10,
color = "salmon", fill = "cornflowerblue",
ggtheme = theme_pander())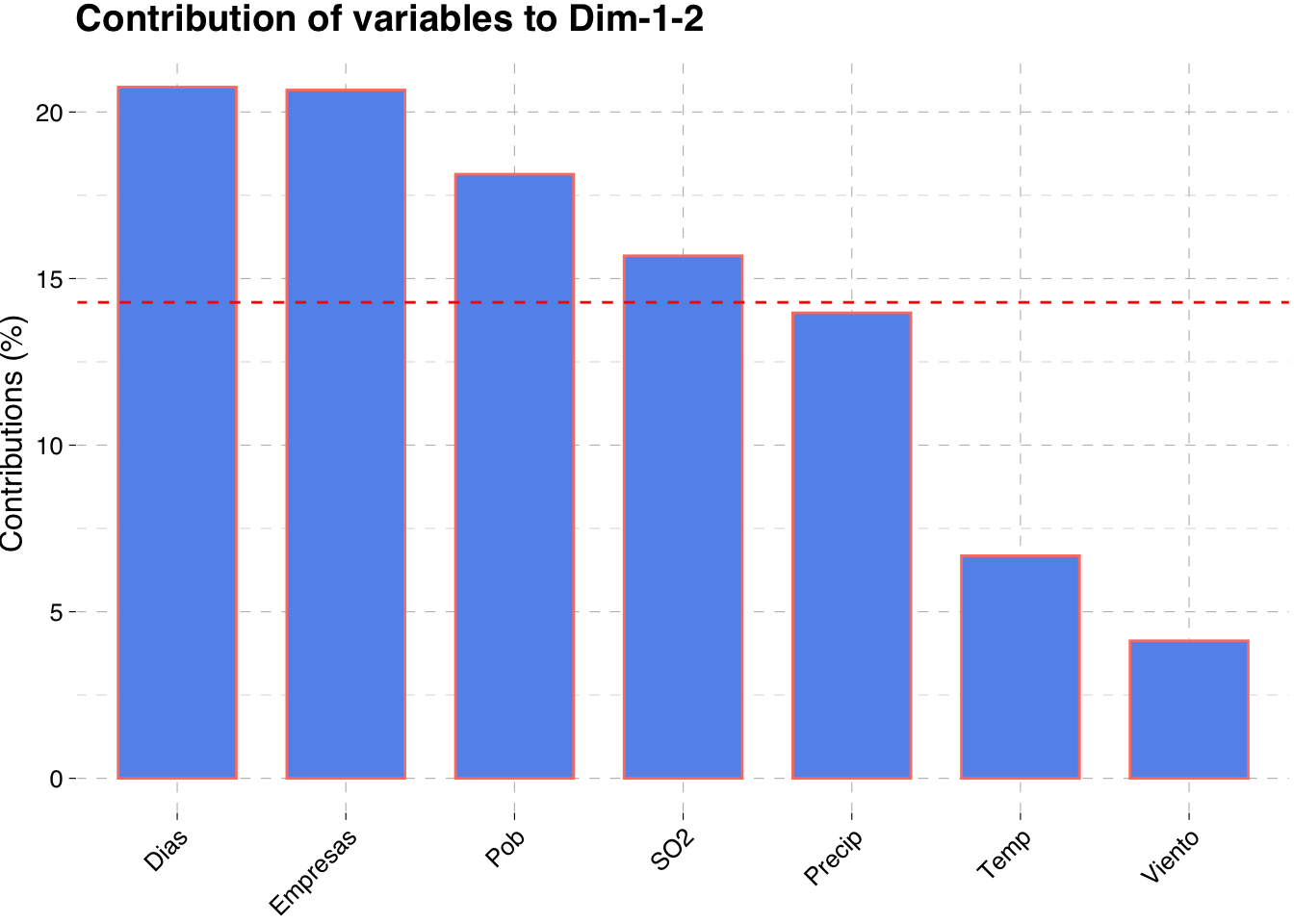
La línea roja discontinua en el gráfico indica la contribución promedio esperada. Si la contribución de las variables fuera uniforme, el valor esperado sería 1/longitud (variables) = Temp y Viento no contribuyen mucho a las dos primeras componentes. No obstante en el apartado de selección de componentes se acordó que serían 3 las CP a elegir.
fviz_contrib(pca, choice = "var", axes = 1:3, top = 10,
color = "salmon", fill = "cornflowerblue",
ggtheme = theme_pander())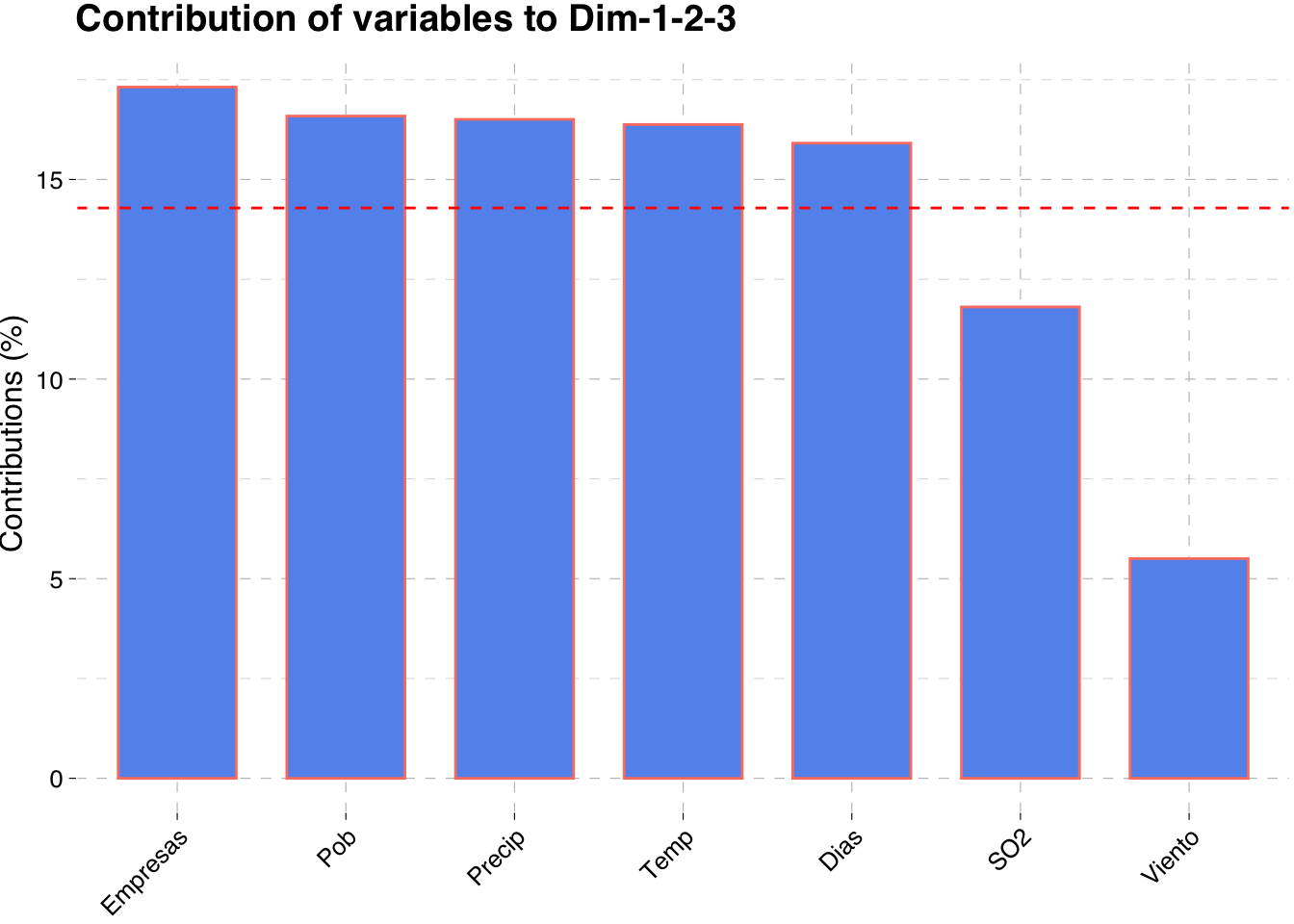
Lo que modifica la interpretación anterior.
Aquí se muestra el círculo de correlación indicando la contribución de las variables a las 2 primeras CP.
fviz_pca_var(pca, col.var = "coord",
gradient.cols = c("orange", "red", "blue"),
ggtheme = theme_pander())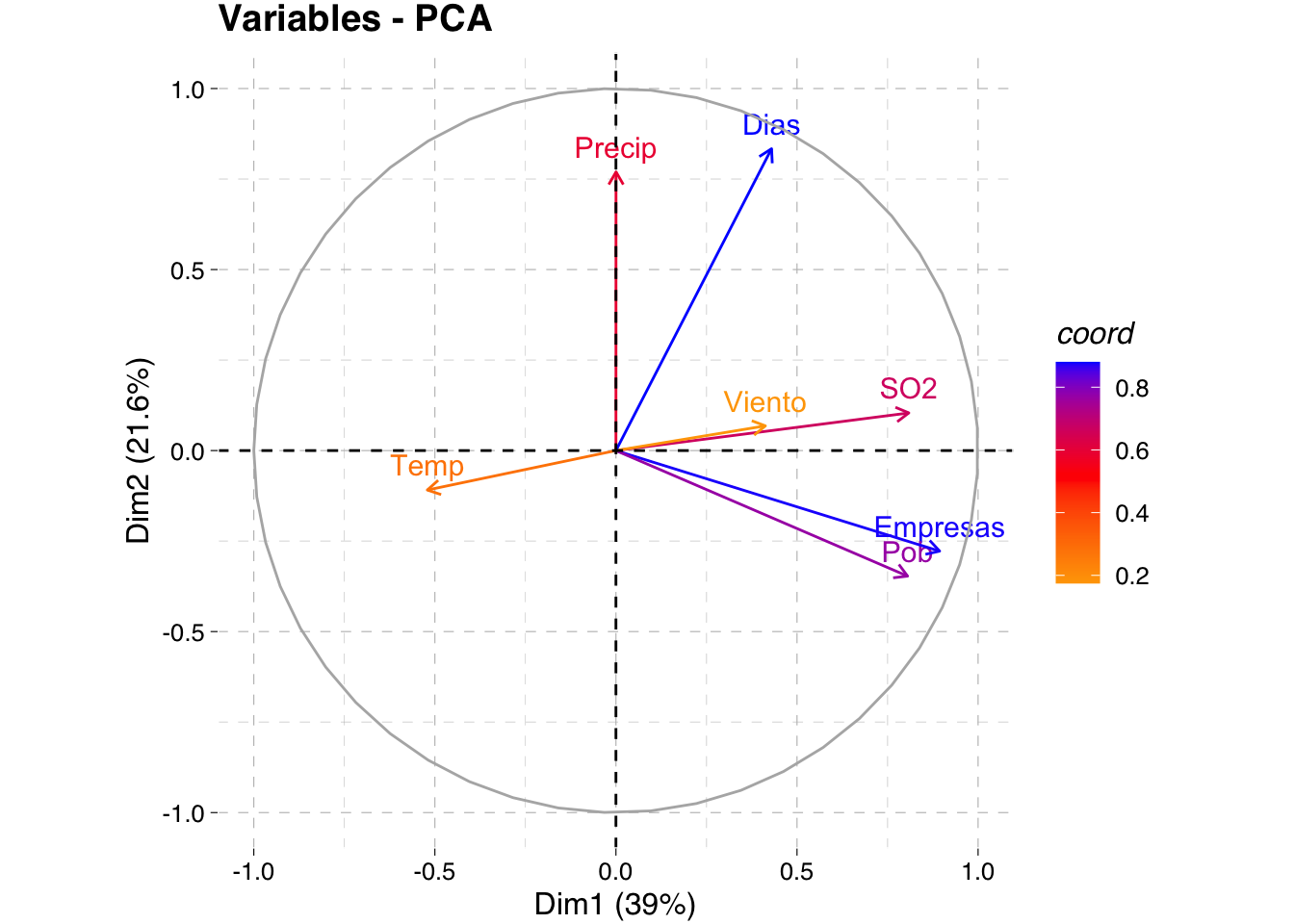
- Gráfico de las puntuaciones
Las puntuaciones son los valores de las CP en las unidades de muestra.
pca$scores %>% head() %>% kable()| Comp.1 | Comp.2 | Comp.3 | Comp.4 | Comp.5 | Comp.6 | Comp.7 | |
|---|---|---|---|---|---|---|---|
| Phoenix | -2.7159758 | -3.8914391 | 1.0583546 | 1.5374428 | 0.0331333 | 0.6544276 | 0.0906066 |
| LittleRock | -1.7177454 | 0.4824200 | 0.8500434 | 0.1945366 | 0.1421327 | -0.5449915 | -0.1986651 |
| SanFrancisco | -0.9389649 | -2.2372464 | -0.1837479 | 0.1527907 | -0.1855924 | -0.2950736 | 0.0071357 |
| Denver | -0.5498615 | -1.9719430 | -1.2285958 | 0.4226922 | -0.3836845 | 0.1301060 | -0.1579881 |
| Hartford | 0.4603718 | 1.0973345 | -0.5897038 | 0.9387046 | 0.7426900 | -0.4004830 | -0.2601886 |
| Wilmington | -0.6970761 | 0.6321489 | -0.4213294 | 0.4888693 | 0.5449854 | -0.2042173 | -0.0507348 |
Podemos representarlas para la CP1 y la CP2. Se podría hacer recurriendo al paquete base de R con plot() o empleando el paquete ggplot, pero aprovechando que se ha introducido el paquete factoextra se usará la función fviz_pca_ind() que permite usar las herramientas de ggplot de forma más intuitiva.
fviz_pca_ind(pca, col.ind = "cornflowerblue",
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"),
repel = TRUE, ggtheme = theme_pander())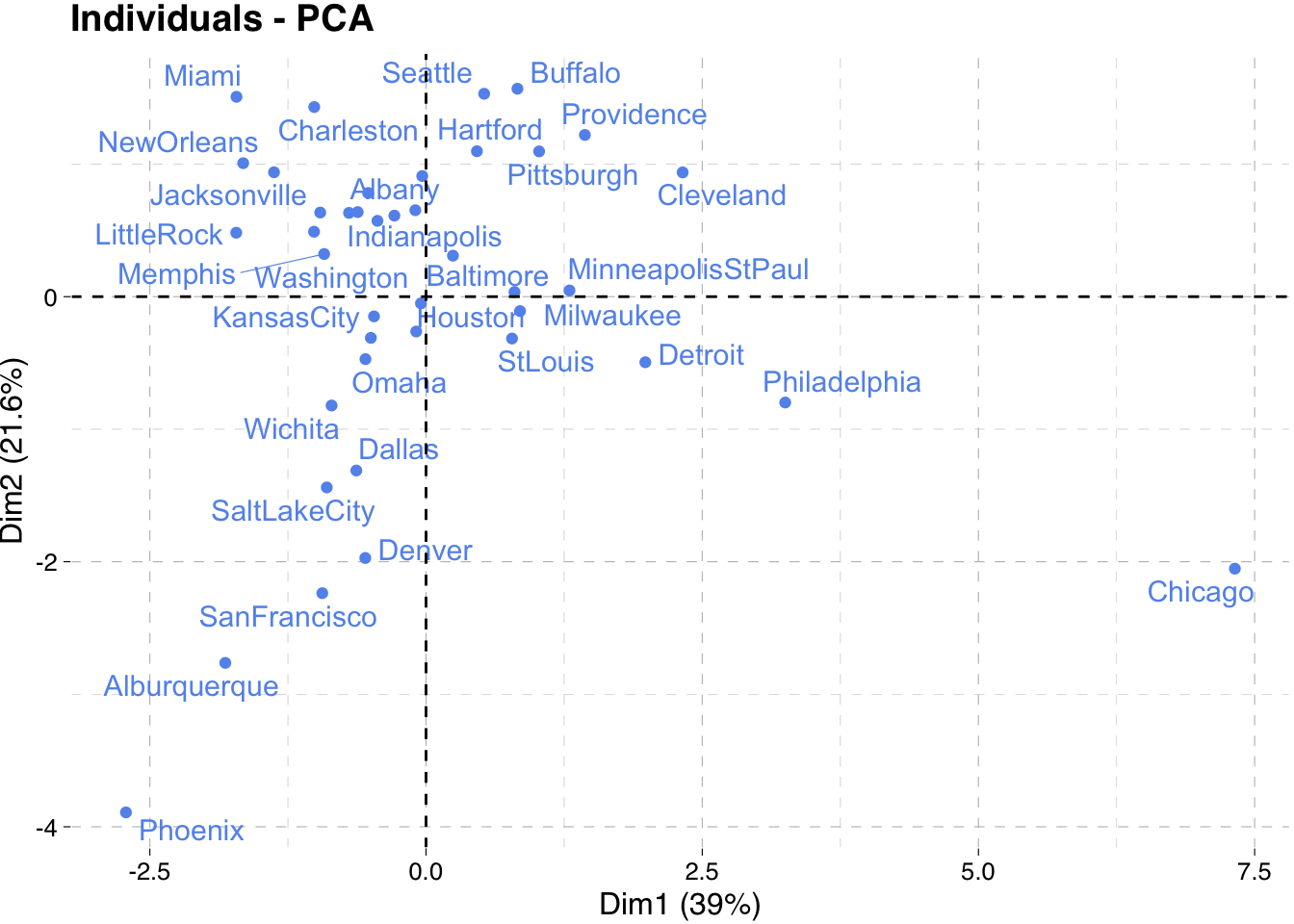
Este gráfico es útil para detectar valores atípicos en el caso multivariado. En este ejemplo, Chicago y Phoenix pueden ser valores atípicos. El gráfico puede verse rotado de nuevo al replicarlo como se explicó anteriormente.
- Biplot
Todo lo anterior se puede agrupar fácilmente en un gráfico conjunto que se conoce como biplot. En el paquete factoextra se encuentra la orden fviz_pca_biplot que permite construir este gráfico de forma intuitiva y haciendo uso del motor de ggplot2.
fviz_pca_biplot(pca,
col.var = "red", # Variables color
col.ind = "cornflowerblue", # Individuals color
repel = TRUE, ggtheme = theme_pander())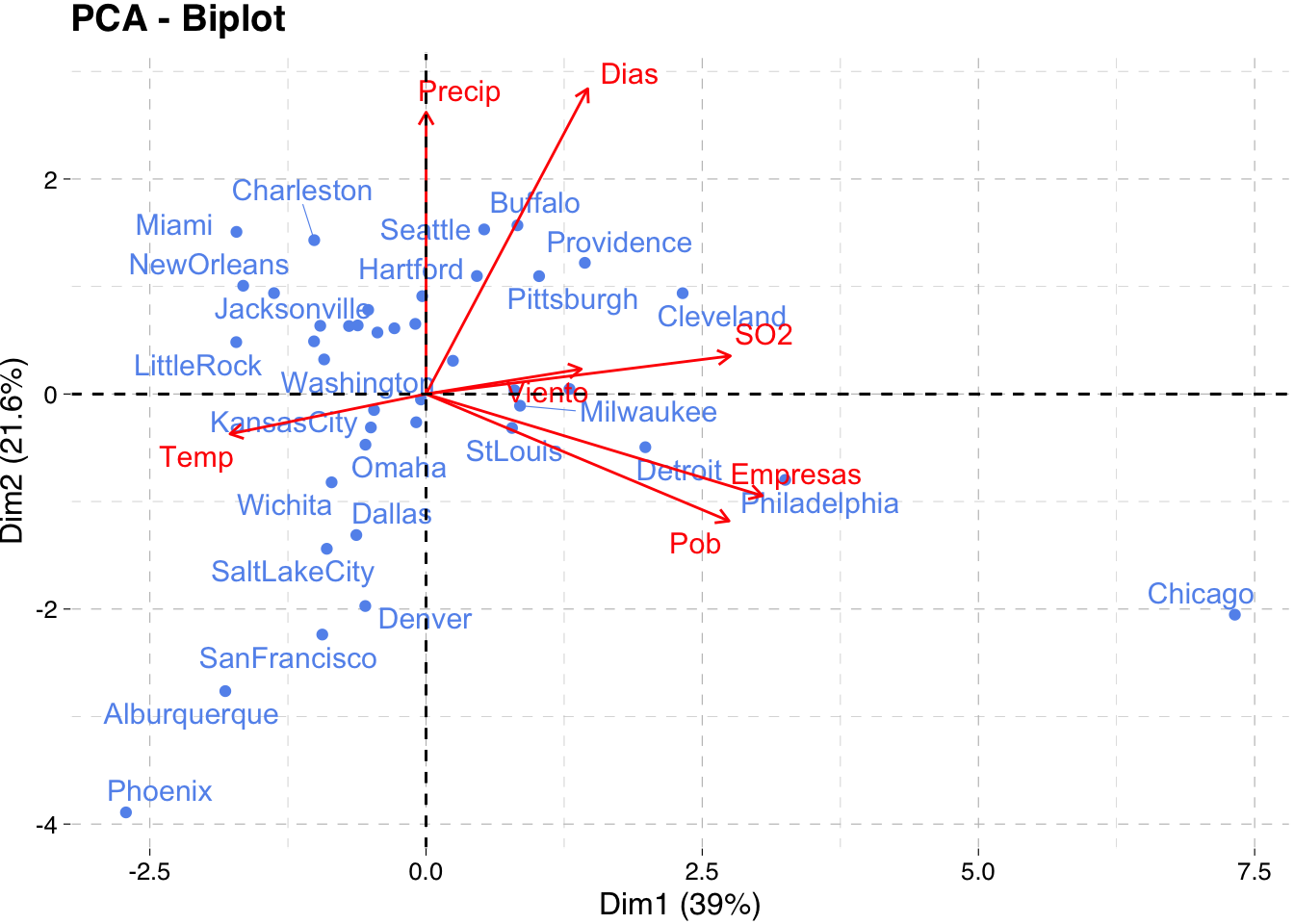
Tenga en cuenta que el biplot solo puede ser útil cuando hay un número bajo de variables e individuos en el conjunto de datos; de lo contrario, la trama final sería ilegible. Tenga en cuenta también que las coordenadas de los individuos y las variables no se construyen en el mismo espacio. Por lo tanto, en el biplot, debe centrarse principalmente en la dirección de las variables, pero no en sus posiciones absolutas en el gráfico.
En términos generales, un biplot se puede interpretar de la siguiente manera:
- Un individuo que está en el mismo lado de una variable dada tiene un valor alto para esta variable.
- Un individuo que está en el lado opuesto de una variable dada tiene un valor bajo para esta variable.
A continuación, comprobamos que la variabilidad de la PC disminuye
pca$scores Comp.1 Comp.2 Comp.3 Comp.4 Comp.5
Phoenix -2.71597577 -3.89143908 1.05835456 1.53744284 0.03313329
LittleRock -1.71774538 0.48242002 0.85004335 0.19453662 0.14213267
SanFrancisco -0.93896495 -2.23724644 -0.18374788 0.15279070 -0.18559238
Denver -0.54986148 -1.97194298 -1.22859584 0.42269224 -0.38368448
Hartford 0.46037184 1.09733455 -0.58970378 0.93870455 0.74269000
Wilmington -0.69707612 0.63214894 -0.42132942 0.48886932 0.54498541
Washington -0.04612550 -0.05089780 0.35423200 -0.04402514 -0.02934285
Jacksonville -1.37601389 0.93839689 1.86596320 -0.45438597 0.01384513
Miami -1.71572800 1.50799472 2.58526278 -0.82939956 0.05685634
Atlanta -0.61858862 0.63808943 0.98836295 -0.19607655 0.11214859
Comp.6 Comp.7
Phoenix 0.6544276475 0.090606552
LittleRock -0.5449915353 -0.198665115
SanFrancisco -0.2950735876 0.007135691
Denver 0.1301060178 -0.157988070
Hartford -0.4004830221 -0.260188627
Wilmington -0.2042172800 -0.050734764
Washington -0.0696776892 0.191097207
Jacksonville 0.1205695112 0.179974967
Miami 0.7195496265 -0.183303486
Atlanta -0.0989937572 -0.071121250
[ reached getOption("max.print") -- omitted 31 rows ]pca$scores %>%
as.tibble() %>%
pivot_longer(everything(),names_to = "item",values_to = "valor") %>%
ggplot(aes(x=item, y=valor, fill=item)) +
geom_boxplot() + theme_pander() +
theme(legend.position = "none") +
xlab("") + ylab("")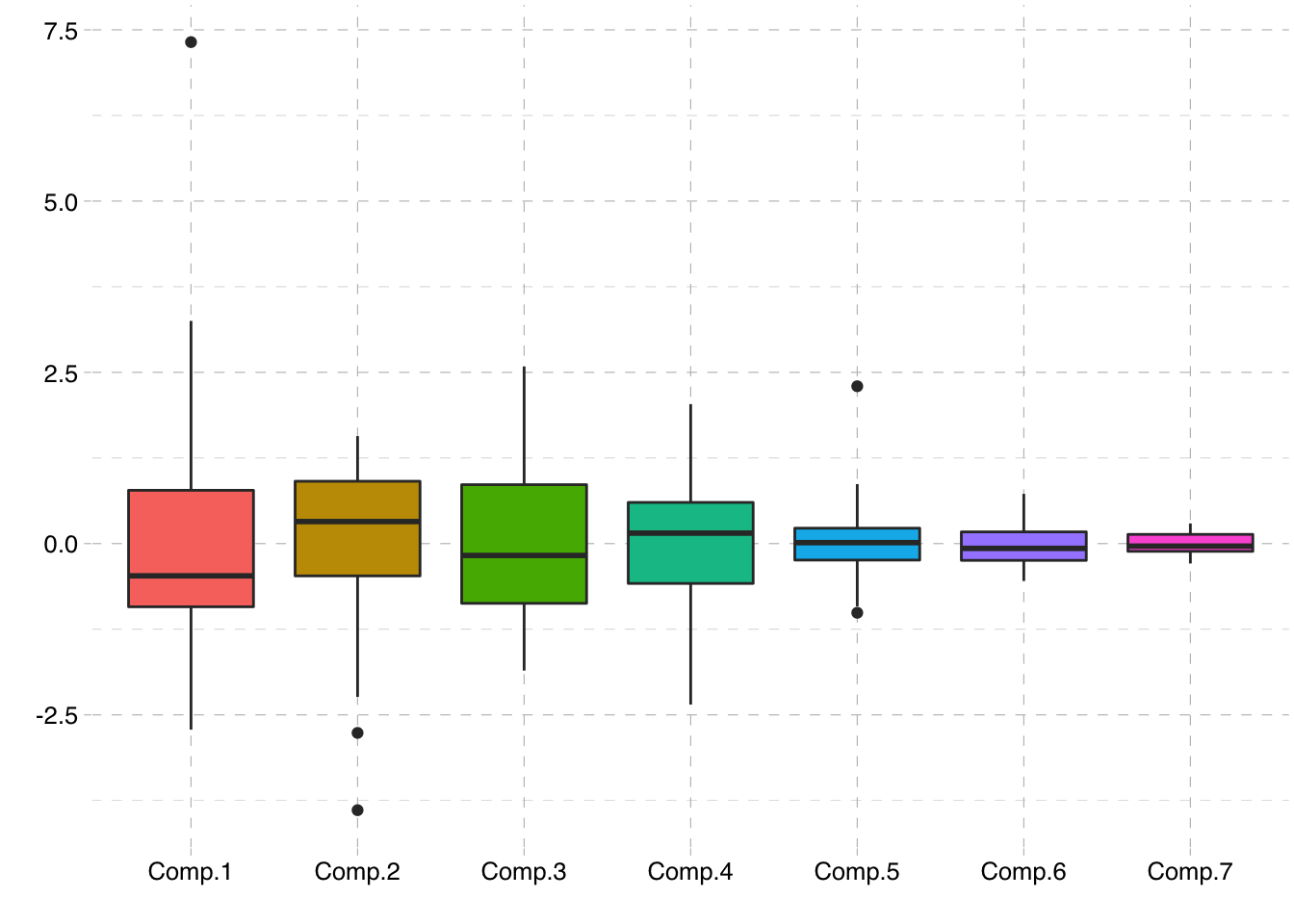
Podemos usar el comando boxplot para detectar los valores atípicos en nuestros datos.
boxPCA = boxplot(pca$scores)outliers = boxPCA$out
outliers[1] 7.320800 -3.891439 -2.763310 -1.009775 2.297055component=boxPCA$group
component[1] 1 2 2 5 5Se puede comprobar que los valores atípicos detectados por CP1 y CP2 corresponden a Chicago, Phoenix y Alburquerque.
Ampliación - Usando tidymodels
library(tidymodels)¿Porque tidymodels?
R es un software de licencia abierta que cuenta con una comunidad activa que es a la vez desarrolladora y explotadora de los paquetes disponibles para su uso. Desde hace un tiempo, un grupo de estos miembros, entre los que cabe destacar a Hadley Wickham o Julia Silge entre otros, llevan centrando sus esfuerzos en organizar y facilitar el uso de R como herramienta creando el tidyverso, mundo ordenado. A esta iniciativa se están uniendo otros muchos desarrolladores que dejan de actualizar sus paquetes para integrarlos en los que se están desarrollando, quedando estos desfasados y en algunos casos desactualizados para nuevas versiones de R.
Esta idea bajo la cual se construye el paquete tidymodels, donde se están recogiendo los principales modelos de aprendizaje e incorporándolos en un flujo de trabajo que facilite su programación y análisis.
Este apartado pretende mostrar los pasos para construir un análisis de componentes principales - PCA - con el paquete tidymodels sobre los datos de USairpollution descritos en el apartado anterior. No nos detendremos tanto en las conclusiones y las interpretaciones de los resultados debido a que se han expuesto en el apartado anterior, así como nos preocuparemos del código necesario.
Seguimos trabajando con los mismos datos.
aire.dat SO2 Temp Empresas Pob Viento Precip Dias
Phoenix 10 70.3 213 582 6.0 7.05 36
LittleRock 13 61.0 91 132 8.2 48.52 100
SanFrancisco 12 56.7 453 716 8.7 20.66 67
Denver 17 51.9 454 515 9.0 12.95 86
Hartford 56 49.1 412 158 9.0 43.37 127
Wilmington 36 54.0 80 80 9.0 40.25 114
Washington 29 57.3 434 757 9.3 38.89 111
Jacksonville 14 68.4 136 529 8.8 54.47 116
Miami 10 75.5 207 335 9.0 59.80 128
Atlanta 24 61.5 368 497 9.1 48.34 115
[ reached 'max' / getOption("max.print") -- omitted 31 rows ]En primer lugar, para usar tidymodels, debemos definir un pipeline, es decir, una serie de pasos de análisis que queremos realizar. Para ello empleamos el paquete R recipes.
pca_recipe <- recipe(~., data = aire.dat)Como el ACP es un algoritmo de aprendizaje no supervisado no habrá que realizar la partición del conjunto de datos característico de los modelos de aprendizaje supervisado. No obstante, si que habrá que preprocesar los datos para adecuarlos al ACP escalando los datos. Y finalmente, especificamos que queremos hacer PCA usando step_pca().
pca_trans <- pca_recipe %>%
step_center(all_numeric()) %>% # center the data
step_scale(all_numeric()) %>% # center the data
step_pca(all_numeric()) # pca on all numeric variables
pca_transData Recipe
Inputs:
role #variables
predictor 7
Operations:
Centering for all_numeric()
Scaling for all_numeric()
No PCA components were extracted.Ahora estamos listos para el análisis. Usaremos la receta que construimos hasta ahora como argumento para la función prep(), “preparar”.
pca_estimates <- prep(pca_trans)
pca_estimatesData Recipe
Inputs:
role #variables
predictor 7
Training data contained 41 data points and no missing data.
Operations:
Centering for SO2, Temp, Empresas, Pob, Viento, Precip, Dias [trained]
Scaling for SO2, Temp, Empresas, Pob, Viento, Precip, Dias [trained]
PCA extraction with SO2, Temp, Empresas, Pob, Viento, Precip, Dias [trained]pca_estimates %>% names()[1] "var_info" "term_info" "steps" "template"
[5] "retained" "tr_info" "orig_lvls" "last_term_info"Nuestros resultados de los pasos de la receta de ACP están disponibles en steps. En nuestro caso usamos tres pasos, dos para normalizar los datos y uno para hacer ACP. Por lo tanto, el objeto steps es una lista de tamaño tres, donde los dos primeros elementos contienen detalles sobre nuestros pasos de normalización y el tercer elemento contiene los resultados de ACP. Revisemos el tercer paso.
pca_tidy <- pca_estimates$steps[[3]]
pca_tidy$terms
<list_of<quosure>>
[[1]]
<quosure>
expr: ^all_numeric()
env: 0x7fb3b28d11c0
$role
[1] "predictor"
$trained
[1] TRUE
$num_comp
[1] 5
$threshold
[1] NA
$options
list()
$res
Standard deviations (1, .., p=7):
[1] 1.6517021 1.2297702 1.1810897 0.9444529 0.5888792 0.3166822 0.1597339
Rotation (n x k) = (7 x 7):
PC1 PC2 PC3 PC4 PC5
SO2 0.4896988171 0.08457563 -0.0143502 0.40421007 0.7303942
Temp -0.3153706901 -0.08863789 -0.6771362 -0.18522794 0.1624652
Empresas 0.5411687028 -0.22588109 -0.2671591 -0.02627237 -0.1641011
Pob 0.4875881115 -0.28200380 -0.3448380 -0.11340377 -0.3491048
Viento 0.2498749284 0.05547149 0.3112655 -0.86190131 0.2682549
Precip 0.0001873122 0.62587937 -0.4920363 -0.18393719 0.1605988
Dias 0.2601790729 0.67796741 0.1095789 0.10976070 -0.4399698
PC6 PC7
SO2 -0.18334573 -0.149529278
Temp -0.61066107 0.023664113
Empresas 0.04273352 0.745180920
Pob 0.08786327 -0.649125507
Viento -0.15005378 -0.015765377
Precip 0.55357384 0.010315309
Dias -0.50494668 -0.008217393
$prefix
[1] "PC"
$keep_original_cols
[1] FALSE
$skip
[1] FALSE
$id
[1] "pca_0gtrC"
attr(,"class")
[1] "step_pca" "step" Vemos que las cargas de cada CP quedan guardadas en el argumento res. Si las comparamos con las obtenidas en princomp vemos que coinciden salvo signo de nuevo.
paste("TIDYMODELS")[1] "TIDYMODELS"pca_tidy$res$rotation %>% round(3) PC1 PC2 PC3 PC4 PC5 PC6 PC7
SO2 0.490 0.085 -0.014 0.404 0.730 -0.183 -0.150
Temp -0.315 -0.089 -0.677 -0.185 0.162 -0.611 0.024
Empresas 0.541 -0.226 -0.267 -0.026 -0.164 0.043 0.745
Pob 0.488 -0.282 -0.345 -0.113 -0.349 0.088 -0.649
Viento 0.250 0.055 0.311 -0.862 0.268 -0.150 -0.016
Precip 0.000 0.626 -0.492 -0.184 0.161 0.554 0.010
Dias 0.260 0.678 0.110 0.110 -0.440 -0.505 -0.008paste("PRINCOMP")[1] "PRINCOMP"pca$loadings
Loadings:
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
SO2 0.490 0.404 0.730 0.183 0.150
Temp -0.315 0.677 -0.185 0.162 0.611
Empresas 0.541 -0.226 0.267 -0.164 -0.745
Pob 0.488 -0.282 0.345 -0.113 -0.349 0.649
Viento 0.250 -0.311 -0.862 0.268 0.150
Precip 0.626 0.492 -0.184 0.161 -0.554
Dias 0.260 0.678 -0.110 0.110 -0.440 0.505
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
SS loadings 1.000 1.000 1.000 1.000 1.000 1.000 1.000
Proportion Var 0.143 0.143 0.143 0.143 0.143 0.143 0.143
Cumulative Var 0.143 0.286 0.429 0.571 0.714 0.857 1.000Accediendo al tercer elemento podemos obtener las desviaciones estándar del análisis de ACP y utilizarlas para calcular el porcentaje de variación explicado por cada CP. Para dar la varianza elevamos al cuadrado, que coinciden con los autovalores de
sdev <- pca_estimates$steps[[3]]$res$sdev
percent_variation <- 100*sdev^2 / sum(sdev^2)
var_df <- tibble(CP = paste0("PC",1:length(sdev))) %>%
mutate(Var = sdev^2,
Per_explained = percent_variation,
Cumulative_Per_Explained = cumsum(Per_explained))
var_df %>% kable()| CP | Var | Per_explained | Cumulative_Per_Explained |
|---|---|---|---|
| PC1 | 2.7281197 | 38.9731383 | 38.97314 |
| PC2 | 1.5123349 | 21.6047836 | 60.57792 |
| PC3 | 1.3949730 | 19.9281856 | 80.50611 |
| PC4 | 0.8919913 | 12.7427327 | 93.24884 |
| PC5 | 0.3467787 | 4.9539809 | 98.20282 |
| PC6 | 0.1002876 | 1.4326799 | 99.63550 |
| PC7 | 0.0255149 | 0.3644989 | 100.00000 |
Ahora podemos construir el gráfico el scree plot que muestra y de forma similar el del porcentaje variabilidad explicada por cada CP.
var_df %>%
mutate(CP = fct_inorder(CP)) %>%
ggplot(aes(x = CP, y = Var)) +
geom_bar(stat="identity",width=0.01) +
geom_point() +
geom_hline(yintercept=1, linetype="dashed", color = "red") +
theme_pander() 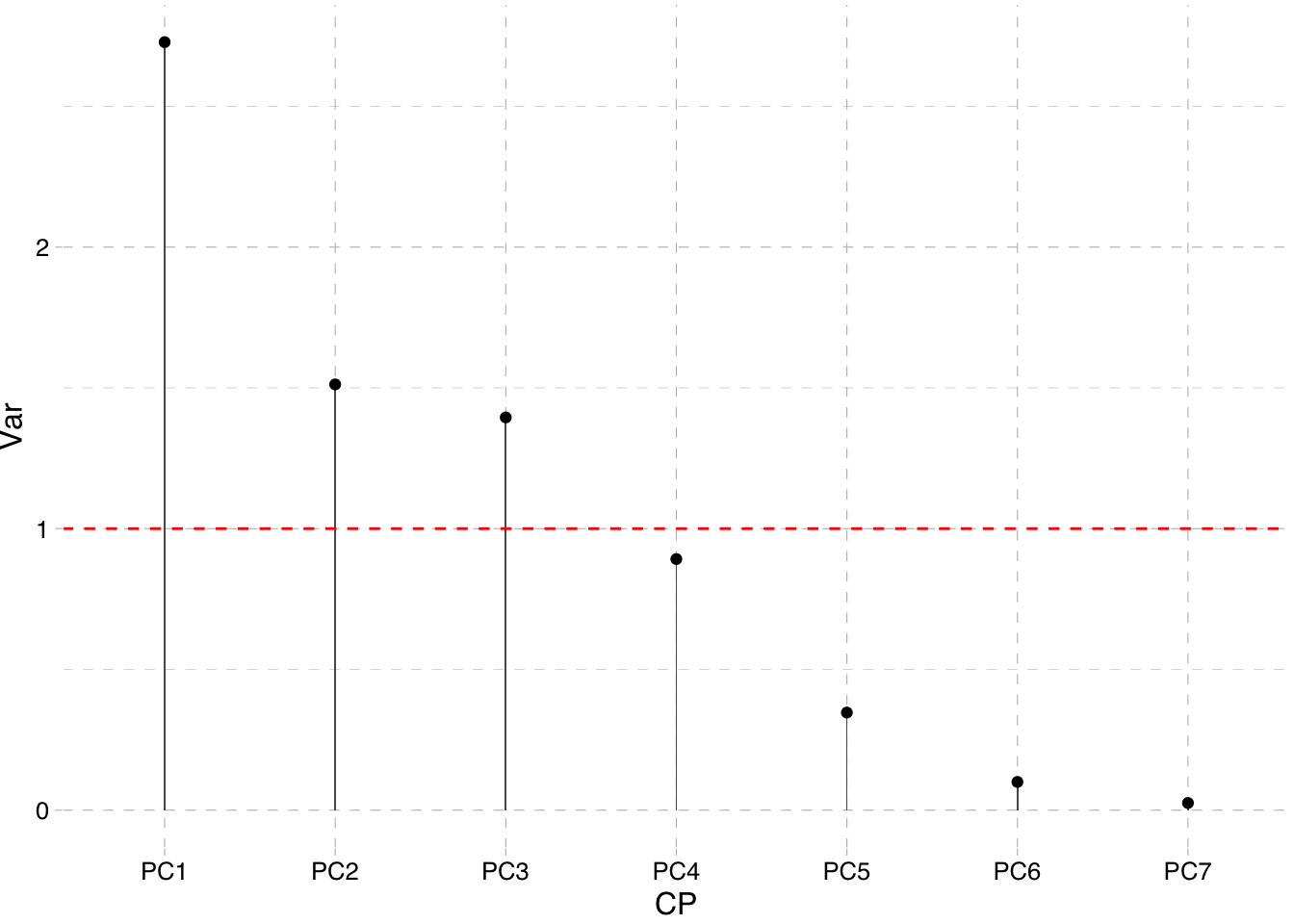
var_df %>%
mutate(CP = fct_inorder(CP)) %>%
ggplot(aes(x = CP, y = Per_explained)) +
geom_col(fill = "cornflowerblue", color = "red") +
ylab("Variablidad explicada") + theme_pander() 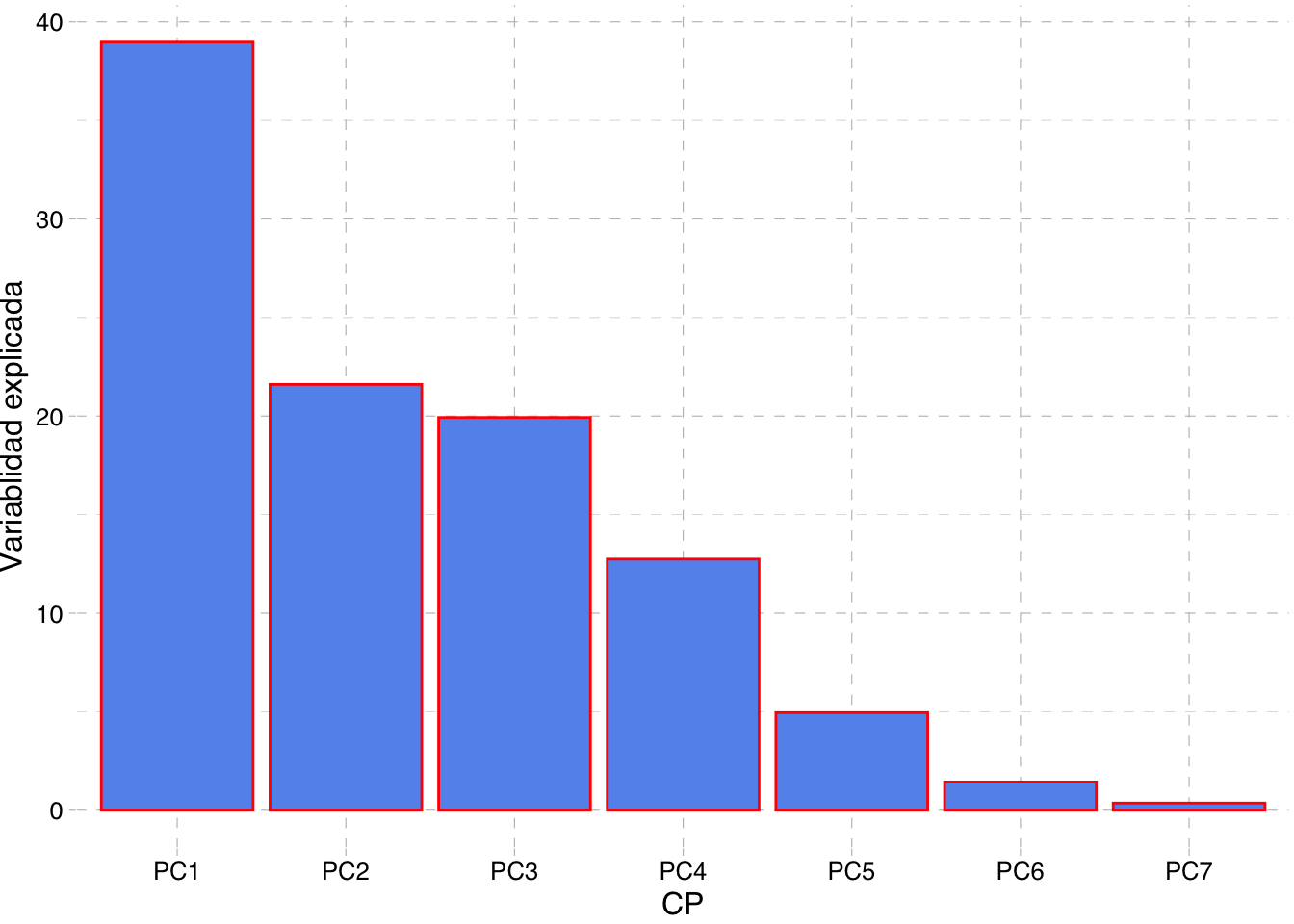
Echemos un vistazo a los componentes principales calculados por nuestra receta. El paquete de recipes de tidymodels tiene una función llamada juice, “jugo”, que devolverá los resultados de una receta creada por prep.
juice(pca_estimates) # A tibble: 41 x 5
PC1 PC2 PC3 PC4 PC5
<dbl> <dbl> <dbl> <dbl> <dbl>
1 -2.68 -3.84 -1.05 1.52 0.0327
2 -1.70 0.477 -0.840 0.192 0.140
3 -0.927 -2.21 0.181 0.151 -0.183
4 -0.543 -1.95 1.21 0.418 -0.379
5 0.455 1.08 0.582 0.927 0.734
6 -0.689 0.624 0.416 0.483 0.538
7 -0.0456 -0.0503 -0.350 -0.0435 -0.0290
8 -1.36 0.927 -1.84 -0.449 0.0137
9 -1.69 1.49 -2.55 -0.819 0.0562
10 -0.611 0.630 -0.976 -0.194 0.111
# … with 31 more rowsPodemos usar los resultados de juice para hacer un diagrama de ACP estándar, un diagrama de dispersión con la CP1 en el eje
juice(pca_estimates) %>%
ggplot(aes(PC1, PC2, label = rownames(aire.dat))) +
geom_point(color="salmon", alpha = 0.7, size = 2,show.legend = FALSE) +
geom_text(check_overlap = TRUE) +
labs(title="PCA with Tidymodels") + theme_pander() 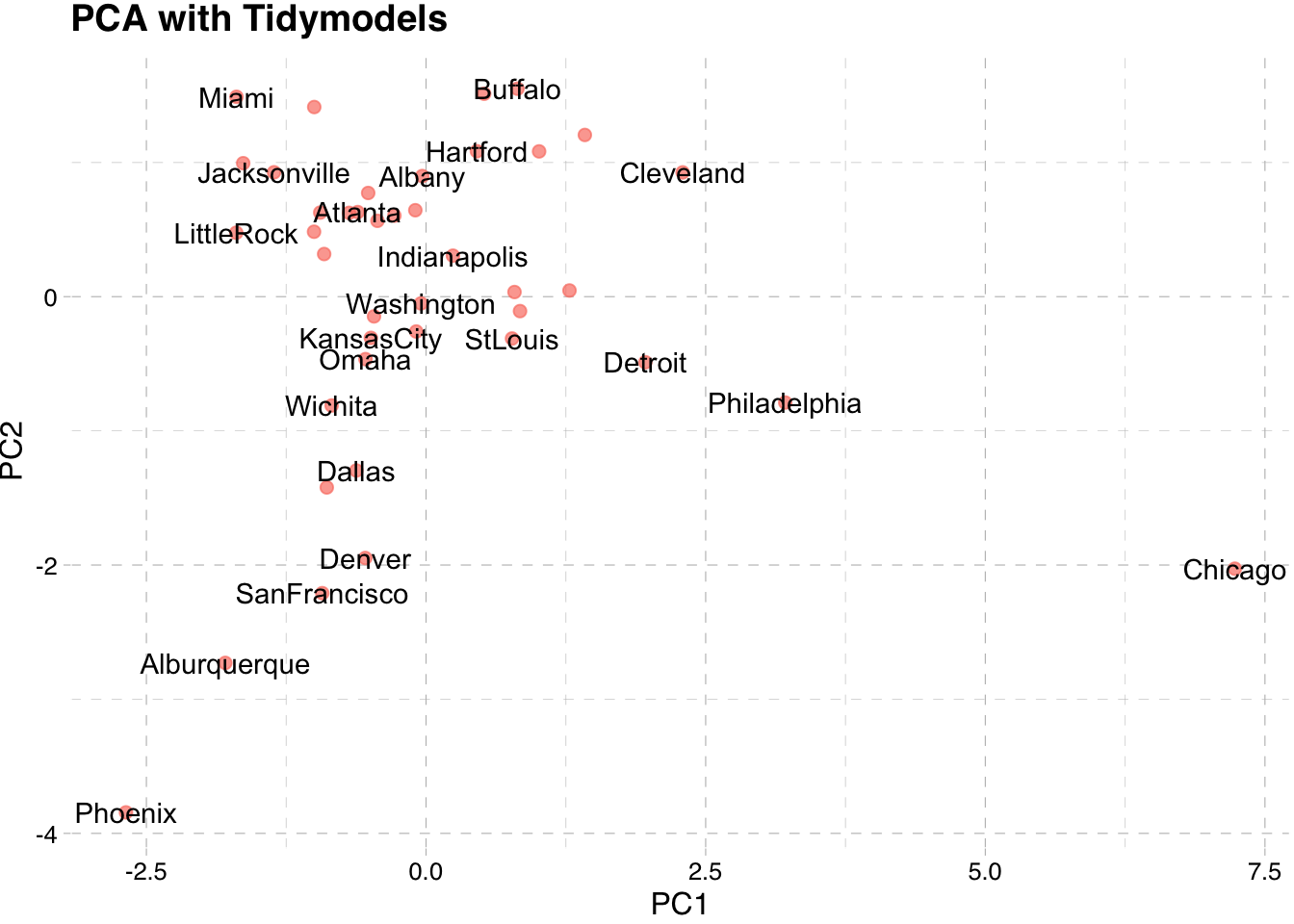
Obviamente el uso de tidymodels supone en ocasiones un mayor esfuerzo en cuanto a código se refiere y que sea una perspectiva novedosa también influye en su comprensión y manejabilidad. No obstante, se sigue un proceso estructurado lo que facilitará su entendimiento a largo plazo y, como se comentaba anteriormente, la comunidad lleva tiempo mostrando interés en metodologías como está que seguramente terminarán imperando en la minería de datos y programación estadística.
Referencias
Making sense of principal component analysis, eigenvectors & eigenvalues
Sobre ACP con tidymodels:
Sobre el signo de las CP:
¿Por qué la interpretación es la misma aunque cambie el signo? El ACP proporciona vectores principales que apuntan en las mejores direcciones para proyectar sus datos en términos de varianza o error al cuadrado, ignorando el signo. Un vector principal que apunta en la dirección opuesta también es una solución válida para ACP pero le dará componentes principales con el signo opuesto. Informalmente, se puede decir que
fix_sign = TRUEalivia eso.↩︎